Histgen_Paper
Histgen
Code:
https://github.com/dddavid4real/HistGen
Data:
Abstract
- 自动生成病理学报告,减轻工作量
- Pathology 是 癌症诊断的黄金标准
- 基于多实例学习并且发布了数据集
- 两个模块
- 局部-全局分层编码器
- 跨模态上下文模块
- 有强大迁移学习能力
Introduction
- Background:
- 数字化WSI得以普及,推动CPATH发展
- WSI Gigabyte 和GPU的限制,MIL被用于WSI分析
- Motivation:
- 撰写报告是劳动密集且容易出错的任务,自动化生成很有前景
- MIL 领域集中在WSI级别的预测,很少有WSI report 生成
- Challenges:
- 缺少数据集,数据集主要关注patch级别的图像和标题,忽略了全面,全局性的描述
- WSI 图像巨大,阻碍现有方法直接使用
- WSI patch 信息密度高(密集的视觉信号) → 简介的诊断报告(离散的文本标记)
- WSI 依赖于MIL,需要pre-train的特征提取器,这是一个关键瓶颈,阻碍了WSI报告的最佳性能
- Contributions:
- 构建了标注数据集
- 提出局部-全局分层视觉编码器
- 跨模态上下文模块
- pre-train一个通用MIL特征提取器
- 通过实验验证了模型的优越性
Method
dataset
- 从TCGA下载诊断报告pdf
- 使用GPT-4进行清洗和摘要
- 将病例ID和WSI进行匹配,形成一个77753样本的WSI-报告数据集
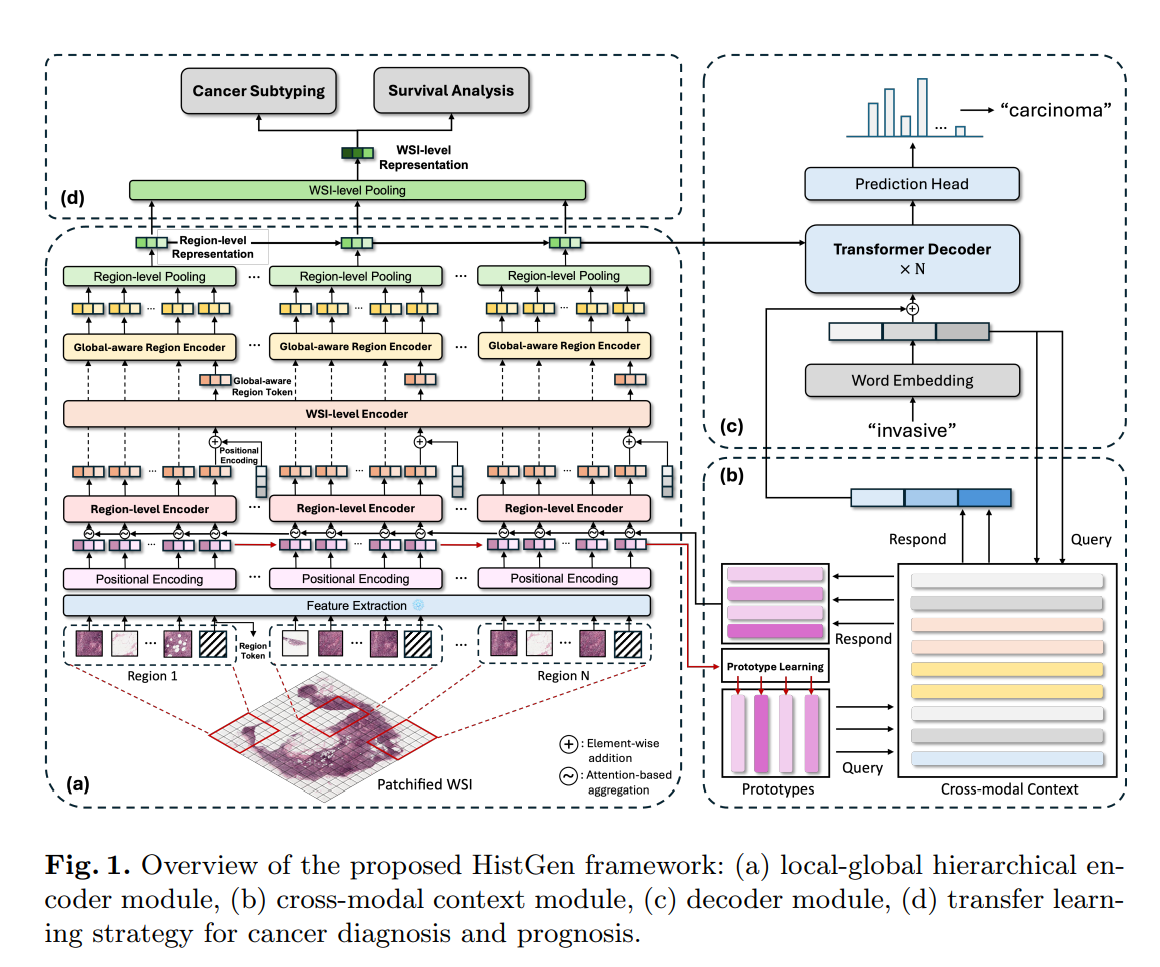
Histgen 框架
- LGH:使用预训练backbone进行提取patch feature,然后从局部到全局方式进行编码
- CMC:链接视觉和文本两种不同的模态,存储迭代中的只是供模型参考
- decoder:生成报告
- transfer learning 模型微调用于WSI级别的下游任务
LGH
- 局部编码,全局交互,信息融合
CMC
- 作为一个外部存贮器,可以被迭代访问和更新
- 对于视觉输入,通过交叉注意力原型学习选取关键补丁作为query,避免序列冗余。CMC生成response,随后聚合会原始视觉特征中,为其注入跨模态信息。这种交互也用哦关于解码器的文本嵌入
- 作为一个外部存贮器,可以被迭代访问和更新
Loss function
- 最大化给定WSI I的情况下生成T的概率
$$
\theta^*=\text{argmax}\theta \sum{i=1}^t\text{log}P(y_i|y_1,y_2,…,y_{i-1},I;\theta)
$$
Experiment & Results
Implementation
- Implementation Details
- Train: test: val = 8:1:1
- 癌症亚型分类:为了测试迁移学习的能力三个外部数据集:UBC-OCEAN、Camelyon和TUPAC16
- 生存分析:在TCGA的六个数据集上进行评估
- Evaluation Metrix:
- NLG:包括BLEU、METEOR和ROUGE-L
- 分类使用蒙特卡洛交叉验证的准确率和AUC
- 生存分析使用蒙特卡洛交叉验证的c-index
- 模型设置
- 特征提取器采用预训练的 DINOOv2 ViT-L,hidden dimension为1024
- LGH模块区域大小为96,解码器为三层的transformer,8个attention head,隐藏状态维度为512
- CMC dimension 512 x 2048
- learning rate 1e-4
- 推理时采用beam search 波束大小为3
- WSI 报告生成结果
- ImageNet预训练的ResNet50、WSI上预训练的CTransPath以及本文的莫i选哪个
- 本文的提升更加显著
- 在所有指标上提升了约3%
- 消融实验
- 以基础模型作为起点(transformer + pooling)
- 叠加LGH和CMC会带来性能的持续提升
- Transfer learning
- 为了证明模型学到了诊断相关信息,将预训练好的模型微调并应用于下游任务
- 癌症亚型分类:
- 显著优于传统方法
- 生存分析:所有6个数据集上保持了最高的平均分
Conclusion
- 核心贡献 引入了 Histgen 一个MIL 框架增强自动化report生成
- 模型设计 local-global 信息,对齐跨模态编码和解码阶段来工作
- 数据集 整理了一个数据集
- 结果 优于传统任务
- 局限性 范围仅限于组织病理学,扩展到其他领域,将跨领域的报告生成视为一个同一问题
Appendix
Discussion
All articles on this blog are licensed under CC BY-NC-SA 3.0 CN unless otherwise stated.